データをクラスタリングするk-means法を書いてみた (Python 編)
以前に MATLAB で書いた教師無しクラスタリング法 k-means のコードを、Python で書き直してみました。ほぼ初の Python コードなので、怪しいかもしれませんが、ひとまず動きました。
↓ 初期状態
↓ 1サイクル目
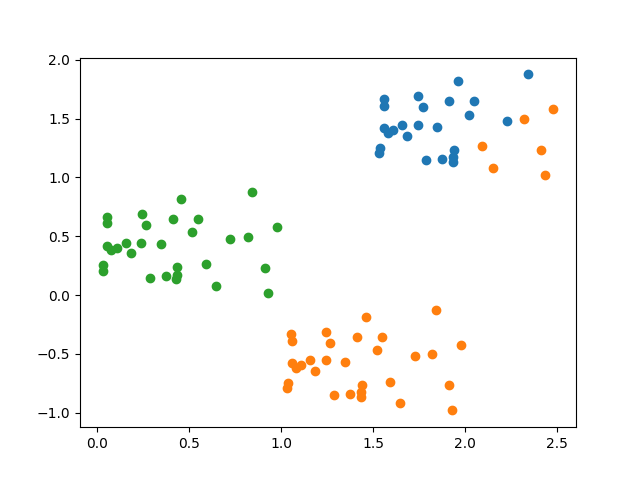
↓ 2サイクル目 (収束)
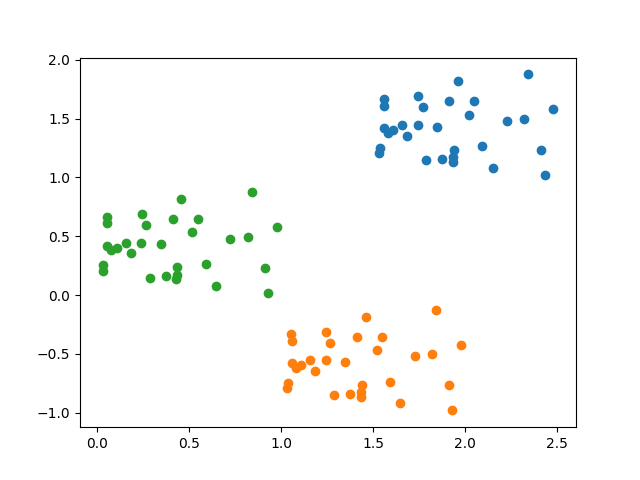
import numpy as np import matplotlib.pyplot as plt def plotPoints(x, y, clu, k): fig = plt.figure() ax = fig.add_subplot(111) for i in range(k): ax.scatter(x[clu == i], y[clu == i]) # data n = 30 x = np.random.rand(n) x = np.hstack([x, x + 1, x + 1.5]) y = np.random.rand(n) y = np.hstack([y, y - 1, y + 1]) # number of clusters k = 3 # assign initial clusters randomly clu = np.random.randint(0, k, len(x)) plotPoints(x, y, clu, k) plt.savefig('0.png') plt.show() ite = 0 while ite < 10: ite += 1 # centroids of clusters vx = [] vy = [] for i in range(k): idx = clu == i vx.append(np.mean(x[idx])) vy.append(np.mean(y[idx])) # distance between the centroid and each point d = np.zeros([n * 3, k]) for i in range(k): d[:, i] = np.sqrt((x - vx[i]) ** 2 + (y - vy[i]) ** 2) # assign new clusters newClu = np.argmin(d, axis=1) if all(newClu == clu): break else: clu = newClu plotPoints(x, y, clu, k) plt.savefig(f'{ite}.png') plt.show()
PyCharm 導入
非線形次元削減法 t-SNE で手書き文字を分類してみた
t-SNE を使って、手書き文字データ MNIST を分類してみました。
MNIST データセットはここから拝借しました。
github.com
MNIST の解説はこれが分かりやすいですね ↓
MNIST データを読みこんで、
% MNIST data load('mnist.mat') % data to be analyzed idx = 1:10000; X = double(testX(idx,:)); c = testY(idx);
t-SNE に放り込んで、ついでに k-means クラスタリングして、
% t-SNE Y = tsne(X,'NumPCAComponents',30); % k-means clustering k = 10; clu = kmeans(Y,k);
プロットしてみました。
subplot(121) scatter(Y(:,1),Y(:,2),5,c,'filled') colorbar colormap jet title('t-SNE on MNIST') subplot(122) scatter(Y(:,1),Y(:,2),5,clu-1,'filled') colorbar colormap jet title('k-means clustering')
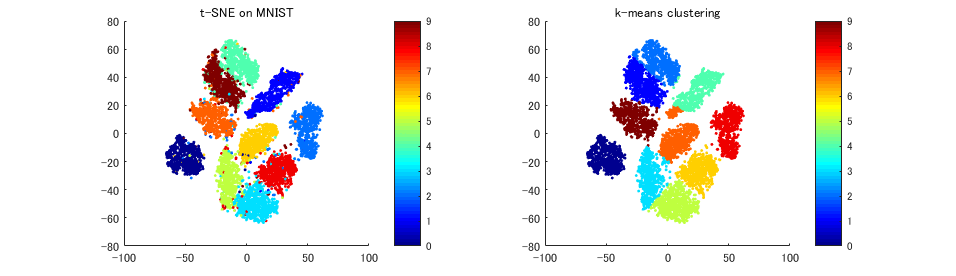
ざっくり適当に放り込んだだけで、ほぼ完璧に分類できています (左図)。t-SNE スゴイ。
k-means でのクラスタリングも良い感じですが (右図)、ところどころ境界がおかしいですね。k-means はクラスタを球形にとってくるので、これは仕方ないのかもしれません。DBSCAN とかにすればもっと上手くいく、かもしれません。
非線形次元削減法 t-SNE でスイスロールを開いてみた
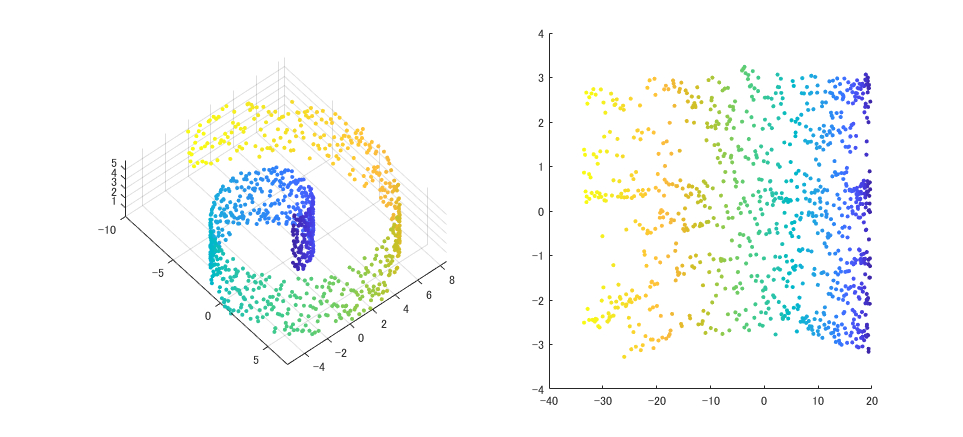
3 次元のスイスロールを、t-SNE を使って開いて (?) みました。MATLAB で書いています。
スイスロールデータを用意し、
% swiss roll data npt= 1000; t = linspace(0,2*pi*1.5,npt); r = linspace(1,10,npt); x = r .* cos(t); y = r .* sin(t); z = rand(1,npt)*10; c = linspace(0,1,npt);
MATLAB の t-SNE 関数に放り込み、
% t-SNE X = [x' y' z']; Y = tsne(X);
プロットしました。
% plot figure(1); clf subplot(121) scatter3(x,y,z,10,c,'filled') axis equal; view([50,50]) title('Original swiss roll') subplot(122) scatter(Y(:,1),Y(:,2),10,c,'filled') title('t-SNE on swiss roll')
開けたのか微妙な感じではありますが、tsne 関数が用意されているので実装は簡単ですね。

Isomap で開いてみた例はこちら ↓
非線形次元削減法 Isomap でスイスロールを開いてみた
多様体学習 (manifold learning) による非線形次元削減法 Isomap を使って、スイスロール風の 3 次元データを開いて 2 次元にしてみました。MATLB で書いています。
自分で適当にスイスロールを作り、
% swiss roll data npt= 1000; t = linspace(0,2*pi*1.5,npt); r = linspace(1,10,npt); x = r .* cos(t); y = r .* sin(t); z = rand(1,npt)*6; c = linspace(0,1,npt); % plot swiss roll figure(1); clf subplot(121) scatter3(x,y,z,10,c,'filled') axis equal; view([50,50])
距離行列 (distance matrix) を計算し、
% distance matrix X = [x' y' z']; D = pdist2(X,X);
いざ、Isomap に放り込みます。
% Isomap n_fcn = 'k'; n_size = 20; options.dims = 1:5; options.display = 0; [Iso,R,E] = Isomap(D, n_fcn, n_size, options); % plot x2 = Iso.coords{2}(1,:); y2 = Iso.coords{2}(2,:); subplot(122) scatter(x2,y2,10,c,'filled')
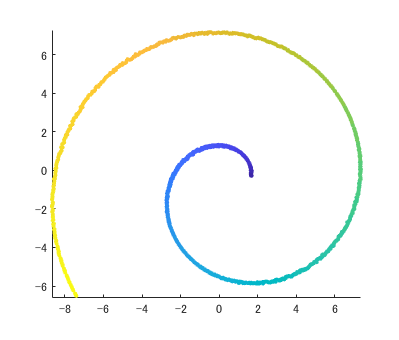
良さそうな感じに開くことができました ↓ 左が元のスイスロール、右が Isomap で開いた図です。n_size は近傍点の数を指定するハイパーパラメータです。これを変えると、見た目やそもそも上手く開けるかが変わります。
非線形次元削減法には、他にも t-SNE や UMAP などいくつもあるので、試してみたいですね。
線形な次元削減法である主成分分析 (PCA) では、ロールを開けません。
[~,score] = pca(X); x3 = score(:,1); y3 = score(:,2); figure(2); clf scatter(x3,y3,10,c,'filled')
Isomap 関数は、こちらのものを使いました ↓
Isomap 関数のなかの L2_distance には、こちらを拝借しました ↓
手書き数字を k-means でクラスタリングしてみた
機械学習で良く使われる手書き数字のデータセット MNIST を使って、k-means 法でどれぐらい教師無しのクラスタリングできるか試してみました (Matlab)。結論から言うと、精度はぜんぜん高くないのですが (正答率 6 割ぐらい)、メモとして残しておきます (構造の違うデータだったらもっとよい精度が出るかも…汗)。
MNIST データセットには、28×28 ピクセルの手書き画像が、訓練用 6 万枚、テスト用 1 万枚、あとこれらの正解ラベルが入っています。
www.atmarkit.co.jp
Matlab のデータファイル形式 (.mat) に成型されたものが下にあったので、それを拝借 (mnist.mat)。
github.com
手順としては、手書き数字データを読み込んで、(そのままだと 28×28 = 784 次元あって計算量が大きいので) 主成分分析 PCA で次元を減らして、k-means でクラスタリング、最後に損失 (= 1-正解率) を計算、という流れです。
まず、MNIST データを読み込んで、uint8 (だと Matlab がエラーを吐くので) を double 型に変換。
load('mnist.mat') trainX = double(trainX); trainY = double(trainY); testX = double(testX); testY = double(testY);
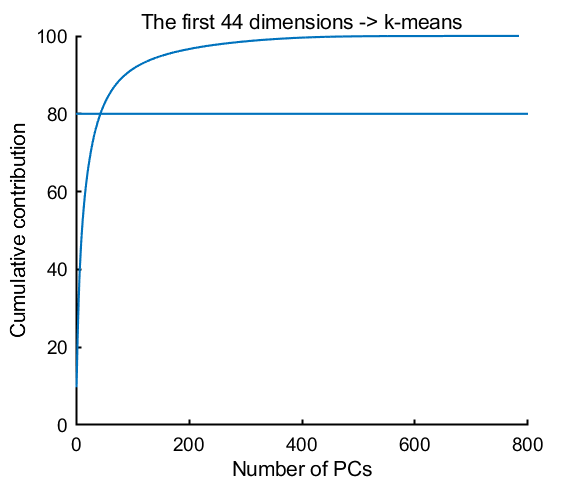
次に、PCA で次元を減らします。寄与率 (explained variance) の累計が 80% になる次元まで使うことにすると、44 次元となりました (784 → 44 に次元削減)。
[~,newX,~,~,explained] = pca(trainX); % explained variance th = 80; cumExplained = cumsum(explained); ndim = find(cumExplained>th,1,'first'); % plot explained variance figure(1); clf plot(cumExplained); refline(0,th) box off xlabel('Number of PCs') ylabel('Cumulative contribution') title(sprintf('The first %u dimensions -> k-means',ndim))
上の 44 次元のデータを k-means でクラスタリングします。k-means はクラスタ数を指定する必要がありますが、0-9 の数字ということで、クラスタ数 10 を指定。
rng(1); % for reproducibility kmeansIdx = kmeans(newX(:,1:ndim),10,'Display','iter','MaxIter',1000);
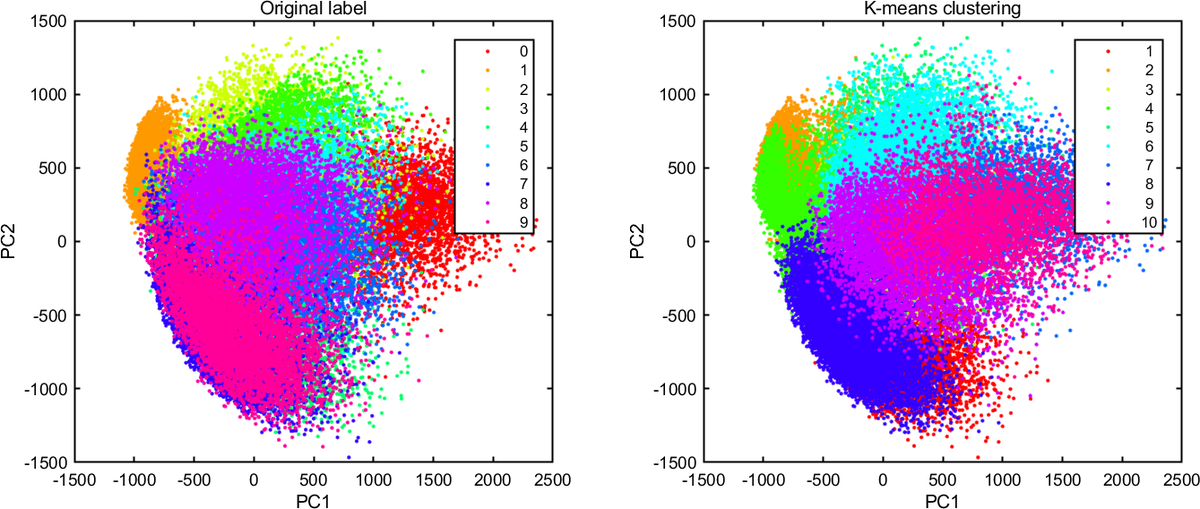
第 2 主成分までプロットしてみて、クラスタリング結果の雰囲気をつかみます。左側は正解ラベルで、右側は k-means のクラスタリング結果です。なんとなく同じように分類できている気もしますが、あからさまに間違えているのもありますね (たとえば、左端の 1 のクラスタ (オレンジ) は、k-means では 2 個のクラスタ (オレンジと緑) に分かれてしまっているように見えます)。
figure(2); clf c = hsv(10); subplot(121); for ii=1:10 plot(newX(trainY==ii-1,1),newX(trainY==ii-1,2),'.','Color',c(ii,:)); hold on end legend(num2str((0:9)')); title('Original label') xlabel('PC1') ylabel('PC2') subplot(122); for ii=1:10 plot(newX(kmeansIdx==ii,1),newX(kmeansIdx==ii,2),'.','Color',c(ii,:)); hold on end legend(num2str((1:10)')); title('K-means clustering') xlabel('PC1') ylabel('PC2')
続いて、クラスタリングの誤答率 (損失) を計算します。k-means の結果 (kmeansIdx) は、実際の 0-9 の数字には対応していないので、どのクラスタにどの数字が多かったかを調べて、各クラスタに対応する数字 0-9 を見つけています。それから誤答率を計算。
idxEdge = -0.5:9.5; idxAxis = 0:9; for ii=1:10 idx = kmeansIdx==ii; N = histcounts( trainY(idx), idxEdge); [~,maxIdx] = max(N); cluValue(ii) = idxAxis(maxIdx); end cluIdx = nan(size(kmeansIdx)); for ii=1:10 cluIdx(kmeansIdx==ii) = cluValue(ii); end % loss L = sum(trainY(:)~=cluIdx)/length(trainY);
最後に、クラスタリング結果の一部を表示してみます。誤答率は 41% (正答率 59%)。半分近く間違えていますが、チャンスレベルの正答率 10% よりはかなり良いでしょう (汗)。一部の数字、0, 2, 6 などは結構よさそうな雰囲気です。1 のクラスタ (と 0 のクラスタも) は、上で見たように 2 つ出現してしまっていますね。
figure(3); clf for ii=1:10 subplot(10,1,ii) Xtmp = trainX(kmeansIdx==ii,:); im = []; for jj=1:20 im = [im reshape(Xtmp(jj,:),28,28)']; end imagesc(im) axis image off text(-10,14,num2str(cluValue(ii))) if ii==1 title(sprintf('Loss = %.2f',L)) end end colormap gray

この方法で、k-means に食べさせる次元数をさらに増やしても、精度はこれ以上ほとんど改善しませんでした。精度を上げたければ、もっと高級な手法を使う必要がありそうです。
今回は、Matlab の Statistics and Machine Learning Toolbox に入っている kmeans 関数を使いましたが、自分で k-means のアルゴリズムを書き下してみた記事はこちら ↓
相互情報量を計算してみた
「脳の情報表現」という本にある「相互情報量 (mutual information)」を計算してみました。
エントロピーは、ある確率変数 X のもつ不確かさを表す量で、下のように定義されます。
2変数の同時確率の場合は、
です。そして、相互情報量は、
と定義されます。この相互情報量という値は、一方の確率変数 X1 の値を知ったときに、もうひとつの確率変数 X2 についての不確かさの減少分を指しています。
この本の20ページ目にある下の例について、実際に計算してみました。
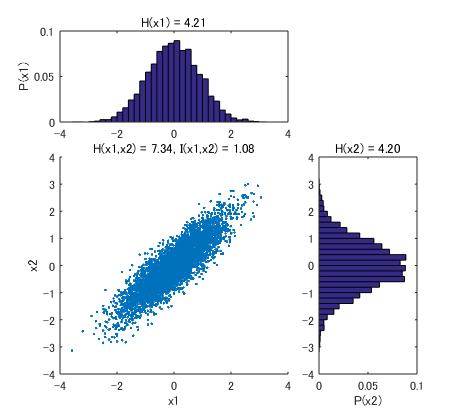
X1 と X2 が相関している場合。相互情報量 I(X1,X2) は 1.08 ビットとなりました (片方の X1 の値を知ると、1.08 ビット分だけ、X2 の値の不確かさが減る)。
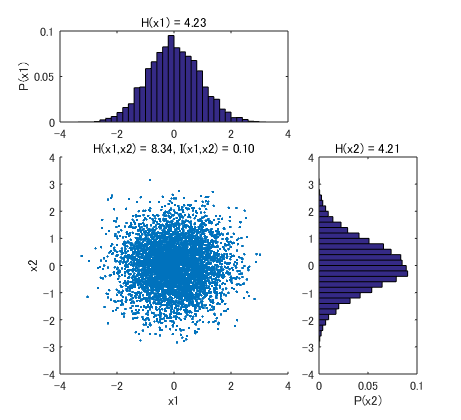
X1, X2 の分布は上と同じですが、片方をシャッフルして独立にした場合。相互情報量は低下して、0.10 ビットとなりました (片方の X1 の値を知っても、もう一方の X2 についての情報はほとんど得られない)。
Matlab コードは下の通りです。
function mi() xaxis = -3.9:0.2:3.9; nbin = numel(xaxis); bin = xaxis(2)-xaxis(1); % 2d gaussian random variables X = mvnrnd([0 0],[0.8 0.7; 0.7 0.8],5000); % shuffling if 0 idx = randperm(size(X,1)); X = [X(:,1) X(idx,2)]; end % probability distribution function pdf1 = hist(X(:,1),xaxis); pdf1 = pdf1/sum(pdf1); pdf2 = hist(X(:,2),xaxis); pdf2 = pdf2/sum(pdf2); pdf12 = zeros(nbin,nbin); for ii=1:nbin for jj=1:nbin pdf12(ii,jj) = sum(xaxis(ii)-bin/2<X(:,1) & X(:,1)<=xaxis(ii)+bin/2 &... xaxis(jj)-bin/2<X(:,2) & X(:,2)<=xaxis(jj)+bin/2); end end pdf12 = pdf12./sum(sum(pdf12)); % entoropy H1 = getEntropy(pdf1); H2 = getEntropy(pdf2); H12= getEntropy(pdf12); % mutual information I12 = H1+H2-H12; % plot figure(1); clf subplot(3,3,[4 8]) scatter(X(:,1),X(:,2),'.') axis([-4 4 -4 4]) title(sprintf('H(x1,x2) = %.2f, I(x1,x2) = %.2f',H12,I12)) xlabel('x1'); ylabel('x2') subplot(3,3,[1 2]) bar(xaxis,pdf1,1) xlim([-4 4]) title(sprintf('H(x1) = %.2f',H1)) ylabel('P(x1)') subplot(3,3,[6 9]) barh(xaxis,pdf2,1) ylim([-4 4]) title(sprintf('H(x2) = %.2f',H2)) xlabel('P(x2)') end function H = getEntropy(pdf) % linearlize pdf = pdf(:); % normalize (just in case) pdf = pdf./sum(pdf); % remove 0 pdf(pdf==0) = []; % entropy H = sum(-pdf.*log2(pdf)); end
